扩展连通性指纹(ECFP/Morgan Fingerprint)介绍
Comment扩展连通性指纹(Extended Connectivity Fingerprints,ECFP)是当下最广受使用用于构建化合物
定量构效关系(QSAR)模型的分子指纹。在 RDKit 中,ECFP 指纹又叫 Morgan 指纹,正因为 ECFP
的核心想法来自于 Morgan 算法,该算法可以为每个原子分配一个唯一的识别符,该标识符会经过几轮迭代。
Morgan 算法
Morgan 算法名字来源于是1965年 Morgan 发表的论文,介绍了 Morgan 算法。该算法可分为两步:
第一步是枚举所有可能的原子位次,其算法如下:
- 随机选择一个原子并分配一个位次,即 1
- 与该原子相邻(成键,不包括氢键)的原子位次依次上涨计数,即 2,3等。
当所有的“原子1”相邻原子都分配完位次后再选择“原子2”分配其相邻原子,已经分配过的原子不再重复分配 - 循环(2)直到所有原子都分配完。或者当有两个分子(如存在离子键或者混合物时),则循环到所有
与“原子1”直接或间接相连的原子都分配完,再随机从剩下原子中选择一个继续分配并按(2)循环
比如对于这个的例子:
这四个原子按上述图片一样的情况链接,则可能会出现如下 12 种分配方案:

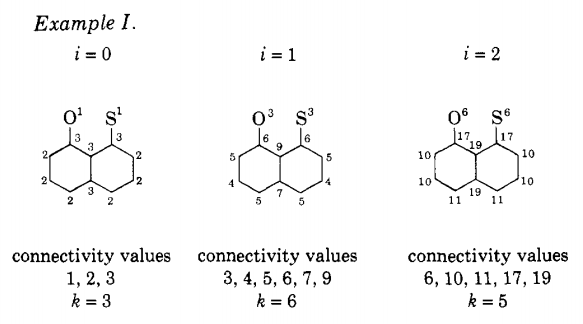
第二步是将上述所有的分配方案进行一一去除,直到只剩最后一种分配方案,则算法结束。在去除时,需要计算每个原子的连通性(Connectivity value),这也是扩展连通性指纹命名的由来。连通性简单的理解就是原子的连接情况,最初等于原子的“度”,即多少根键,然后进行迭代,直到原子多样性达到最大。如下面例子,迭代的算法是新的连通性等于周边原子的(原)连通性之和(感谢在评论中指正)。k 为这些连通性的多样性,即多少个唯一的值,当 k 开始下降则停止迭代。如下图的例子,当 i 为 2 时,发现 k 比原来低,则使用 i=1 的情况。
扩展连通性指纹算法
扩展连通性指纹是一种环形指纹(Circular fingerprint),因为其定义需要设定半径 n(即迭代次数),然后计算每个原子环境识别符(identifier),该识别符类似 Morgan 指纹中的连通性,只是这里的识别符,最终是由半径为 n 的环境决定的。n=1 时称之 ECFP_2,n=2 时为 ECFP_4,以此类推。当 n=0 时,便只考虑原子本身。
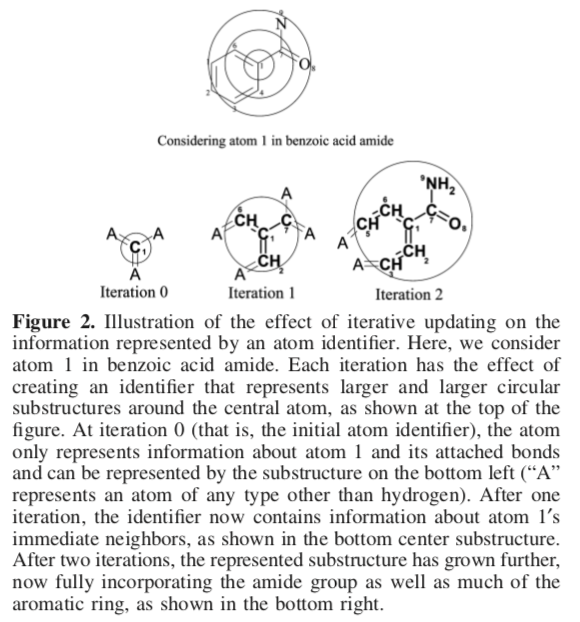
识别符计算有三个阶段:
- 初始化,即给每个原子一个初始标识符,可以是原子序号、原子质量等
- 迭代更新,将该原子周边的原子的信息增加到该原子上,形成数组,然后哈希至一个数字
- 去除冗余,将一些原子环境一致但标识符不同的情况识别出来,进行删除
分子指纹算法如下:
- 创建集合 S 储存所有原子的识别符
- 使用 32 位的整数标记每个原子,比如可以使用 Morgan 算法或者 CANGEN 算法,然后将他们哈希化
- 将每个原子的识别符加入到 S 中
- 当 n=0 时算法结束,否则进入第 5 步
- 对于每个原子,创建一个“键列表”储存该原子周边原子的信息,该列表先根据键级(单键、双键、三建)排序,在根据周边原子识别符大小排序
- 用如下信息填充上述列表:内容为 [n, indentifier, bo1, aid1, bo2, aid2, …],其中 n 为迭代次数,开始为0,bo1 为第 1 根键的键级,aid1 是第 1 根键所连原子的识别符
- 计算第 6 步中的特征列表的哈希值,作为该原子新的识别符
- 如果新算出的识别符在结构上与 S 中的不重复,则加入到 S 中
- 如果 n>=2,则重复5的步骤,进入下一个迭代
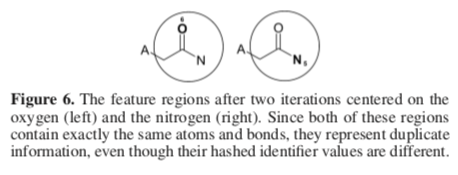
这里所谓结构上不重复,原论文给出了一个例子,从 O 出发和从 N 出发,当 n=2 时,都会出现图中圈出的子结构,由于他们“起始”原子不同,所以标识符肯定也不同,但从结构的角度,他们是一样的,所以需要去除。
官能团类型指纹(FCFP)与 ECFP 异同
FCFP 与 ECFP 类似,都是通过迭代的方式将原子周围环境编译成一个分子指纹,与 ECFP 不同的是初始化时的原子特征。
在 ECFP 中原子的初始识别符类似使用 CANGEN 算法中的方法,即由原子本身的几个性质得到,包括:
- 原子连接数
- 非氢化学键数
- 原子序号
- 原子电荷正负
- 原子电荷绝对值
- 连接氢原子个数
RDKit 中具体实现方法仅仅是用了“1, 3, 5, 6”,代码 详见此处。1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24void getConnectivityInvariants(const ROMol &mol, std::vector<uint32_t> &invars,
bool includeRingMembership) {
unsigned int nAtoms = mol.getNumAtoms();
PRECONDITION(invars.size() >= nAtoms, "vector too small");
gboost::hash<std::vector<uint32_t>> vectHasher;
for (unsigned int i = 0; i < nAtoms; ++i) {
Atom const *atom = mol.getAtomWithIdx(i);
std::vector<uint32_t> components;
components.push_back(atom->getAtomicNum());
components.push_back(atom->getTotalDegree());
components.push_back(atom->getTotalNumHs());
components.push_back(atom->getFormalCharge());
int deltaMass = static_cast<int>(
atom->getMass() -
PeriodicTable::getTable()->getAtomicWeight(atom->getAtomicNum()));
components.push_back(deltaMass);
if (includeRingMembership &&
atom->getOwningMol().getRingInfo()->numAtomRings(atom->getIdx())) {
components.push_back(1);
}
invars[i] = vectHasher(components);
}
} // end of getConnectivityInvariants()
而 FCFP 则不同,使用“官能团类型”作为原子标识符,此处的官能团更像药物化学中的药效团,包括
- 氢键受体
- 氢键供体
- 可电离成阴离子
- 可电离成阳离子
- 芳香原子
- 卤素原子
RDKit 使用预先定义的 SMARTS(参考文档)来匹配该原子是否满足上述六种性质,其代码 详见此处。1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50const char *smartsPatterns[6] = {
"[$([N;!H0;v3,v4&+1]),\
$([O,S;H1;+0]),\
n&H1&+0]", // Donor
"[$([O,S;H1;v2;!$(*-*=[O,N,P,S])]),\
$([O,S;H0;v2]),\
$([O,S;-]),\
$([N;v3;!$(N-*=[O,N,P,S])]),\
n&H0&+0,\
$([o,s;+0;!$([o,s]:n);!$([o,s]:c:n)])]", // Acceptor
"[a]", // Aromatic
"[F,Cl,Br,I]", // Halogen
"[#7;+,\
$([N;H2&+0][$([C,a]);!$([C,a](=O))]),\
$([N;H1&+0]([$([C,a]);!$([C,a](=O))])[$([C,a]);!$([C,a](=O))]),\
$([N;H0&+0]([C;!$(C(=O))])([C;!$(C(=O))])[C;!$(C(=O))])]", // Basic
"[$([C,S](=[O,S,P])-[O;H1,-1])]" // Acidic
};
void getFeatureInvariants(const ROMol &mol, std::vector<uint32_t> &invars,
std::vector<const ROMol *> *patterns) {
unsigned int nAtoms = mol.getNumAtoms();
PRECONDITION(invars.size() >= nAtoms, "vector too small");
std::vector<const ROMol *> featureMatchers;
if (!patterns) {
featureMatchers.reserve(defaultFeatureSmarts.size());
for (std::vector<std::string>::const_iterator smaIt =
defaultFeatureSmarts.begin();
smaIt != defaultFeatureSmarts.end(); ++smaIt) {
const ROMol *matcher = pattern_flyweight(*smaIt).get().getMatcher();
CHECK_INVARIANT(matcher, "bad smarts");
featureMatchers.push_back(matcher);
}
patterns = &featureMatchers;
}
std::fill(invars.begin(), invars.end(), 0);
for (unsigned int i = 0; i < patterns->size(); ++i) {
unsigned int mask = 1 << i;
std::vector<MatchVectType> matchVect;
// to maintain thread safety, we have to copy the pattern
// molecules:
SubstructMatch(mol, ROMol(*(*patterns)[i], true), matchVect);
for (std::vector<MatchVectType>::const_iterator mvIt = matchVect.begin();
mvIt != matchVect.end(); ++mvIt) {
for (const auto &mIt : *mvIt) {
invars[mIt.second] |= mask;
}
}
}
} // end of getFeatureInvariants()
可见 FCFP 所描述的原子本身信息更少,仅仅是描述原子所处的官能团环境,随着迭代的过程,FCFP 似乎在描述几个官能团之间的拓扑关系,比如 FCFP4 将反映距离为2根键内的药效团组合,如 O=C-Cl 这样。
RDKit 中 ECFP 实现源码解析
具体算法实现 详见此处,这里挑选比较关键的地方:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45void calcFingerprint(const ROMol &mol, unsigned int radius,
std::vector<uint32_t> *invariants,
const std::vector<uint32_t> *fromAtoms, bool useChirality,
bool useBondTypes, bool useCounts,
bool onlyNonzeroInvariants, BitInfoMap *atomsSettingBits,
T &res) {
const ROMol *lmol = &mol;
unsigned int nAtoms = lmol->getNumAtoms();
// 计算初始标识符,更新到 invariants 变量
getConnectivityInvariants(*lmol, *invariants);
// layer就是当前迭代次数,ECFP4 则表示迭代2次,radius为2
for (unsigned int layer = 0; layer < radius; ++layer) {
std::vector<uint32_t> roundInvariants(nAtoms);
// 遍历每个原子(即更新每个原子的标识符)
BOOST_FOREACH (unsigned int atomIdx, atomOrder) {
const Atom *tAtom = lmol->getAtomWithIdx(atomIdx);
boost::tie(beg, end) = lmol->getAtomBonds(tAtom);
std::vector<std::pair<int32_t, uint32_t>> nbrs;
// 获取该原子周围(成键)原子,存入 nbrs
while (beg != end) {
const Bond *bond = mol[*beg];
unsigned int oIdx = bond->getOtherAtomIdx(atomIdx);
bt = static_cast<int32_t>(bond->getBondType());
nbrs.push_back(std::make_pair(bt, (*invariants)[oIdx]));
++beg;
}
// 对周围原子排序,这里排序方式貌似比较粗暴,并不如前文步骤(6)中所言
std::sort(nbrs.begin(), nbrs.end());
std::uint32_t invar = layer;
gboost::hash_combine(invar, (*invariants)[atomIdx]);
// 排序后进行再次遍历,用于计算新的标识符
for (std::vector<std::pair<int32_t, uint32_t>>::const_iterator it =
nbrs.begin();
it != nbrs.end(); ++it) {
// add the contribution to the new invariant:
gboost::hash_combine(invar, *it);
}
// 最后将新的 invar 更新到原子的标识符
roundInvariants[atomIdx] = static_cast<uint32_t>(invar);
// 下面要进行原子环境去重,保证该环境之前未出现过,见步骤(8);此处省略
}
// 更新原子标识符
std::copy(roundInvariants.begin(), roundInvariants.end(),
invariants->begin());
}
仔细看hash的过程,分别是1
2
3
4
5std::uint32_t invar = layer;
gboost::hash_combine(invar, (*invariants)[atomIdx]);
for (it){
gboost::hash_combine(invar, *it);
}
这个过程对应的就是步骤(6)中的hash([n, indentifier, bo1, aid1, bo2, aid2, ...])
但个人感觉还是有点不一样的,在 RDKit 中,hash 的顺序类似于[n, identifier, *it1, *it2],这里的it是一个指针,指向nbrs这个数组,而这个数组里每个值是(bt, (*invariants)[oIdx]),即键的类型以及键所连原子的标识符。
类似于hash([n, identifier, [bo1, aid1], [bo2, aid2], ...])不过反正如果顺序没错,里面内容是一样的,因此即使 hash 结果不同也不影响 RDKit 对 ECF P算法的解读。