使用强化学习生成模型产生新的分子
Comment在药物设计、化学信息学领域,深度学习并不一定能比传统机器学习领域有更好的表现,尤其是在学术界,由于有标签的数据很少,数据噪音较大,往往用深度学习只会导致过拟合降低模型的预测能力。但是,生成学习用于药物设计却完全发挥了当前深度神经网络的优势。虽然有标签的数据很少,但是药物设计数据库诸如CHEMBL不乏质量较高的“化合物信息”。这里说的质量较高指的是他们的类药性较强,往往此类数据库中的化合物都是早期药物设计中已经合成出且具有一定生物活性的分子。这些化合物可以统统拿来构建一个模型用于生成新的小分子。
另一方面,深度学习所推崇的”原始数据”,对于图形数据,序列数据的处理能力远远大于传统的机器学习方法,从而解决了传统方法用分子描述符和分子指纹描述分子并不能得到分子结构本身。现在有了深度学习,一切从分子本身(比如分子的 SMILES)进行训练,生成新的SMILE即得到小分子的结构。
RNN生成模型
生成模型的架构主要是循环神经网络 (RNN),从下图可以看出,在一个 RNN 模型里,最初的输入是“GO”,接下来每个输入都是上个单元期待的输出,最终输出是“EOS”。比如一个乙醇分子 CCO,对应这里的 x1, x2, x3。训练的目的就是让x1=C, x2=C, x3=O的概率之积最高,用损失函数表示就是:
$$ J(Θ) = -\sum_{t=1}^{T}logP(X^t|x^{t-1},…,x^1) $$
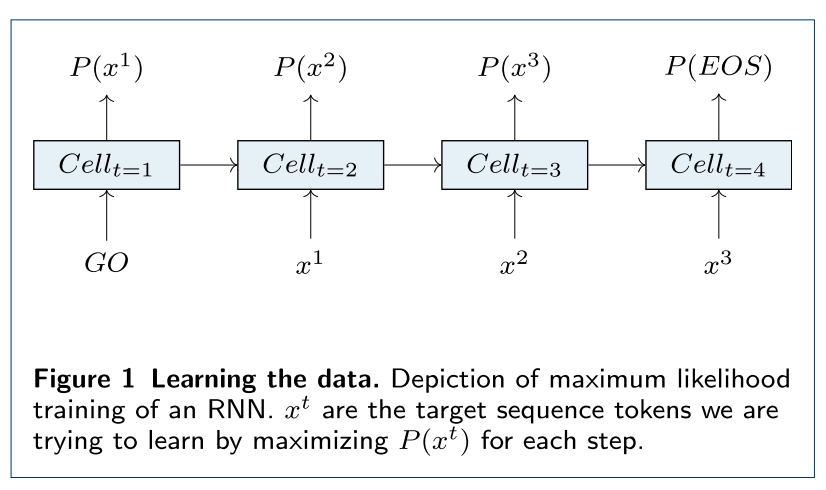
当需要产生新的分子时,则只要让输入时“GO”,然后让前一个单元的输出作为后一个单元的输入,直到有一个单元输出“EOS”表示完成一条数据的生成。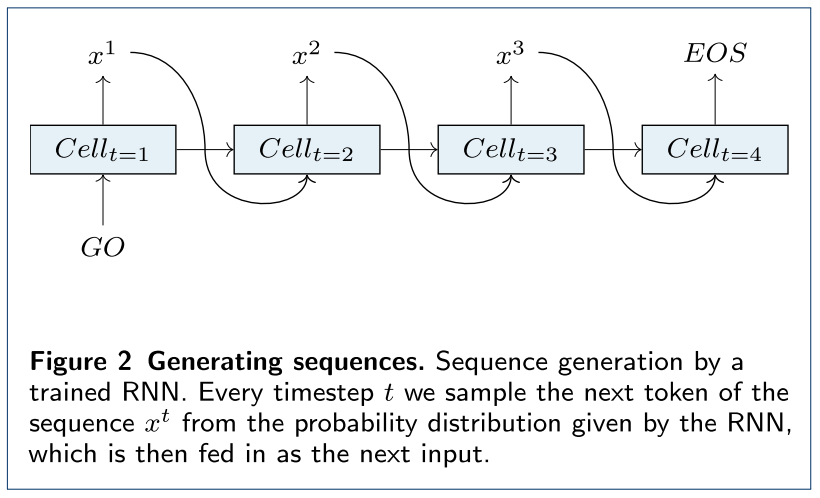
RL优化生成的化合物
当使用大量数据训练RNN生成模型后,该模型(称之为 Prior)具有生成 SMILES 的能力,接下来是通过强化学习来优化这个模型(Agent),让其能生成“更好的分子”。
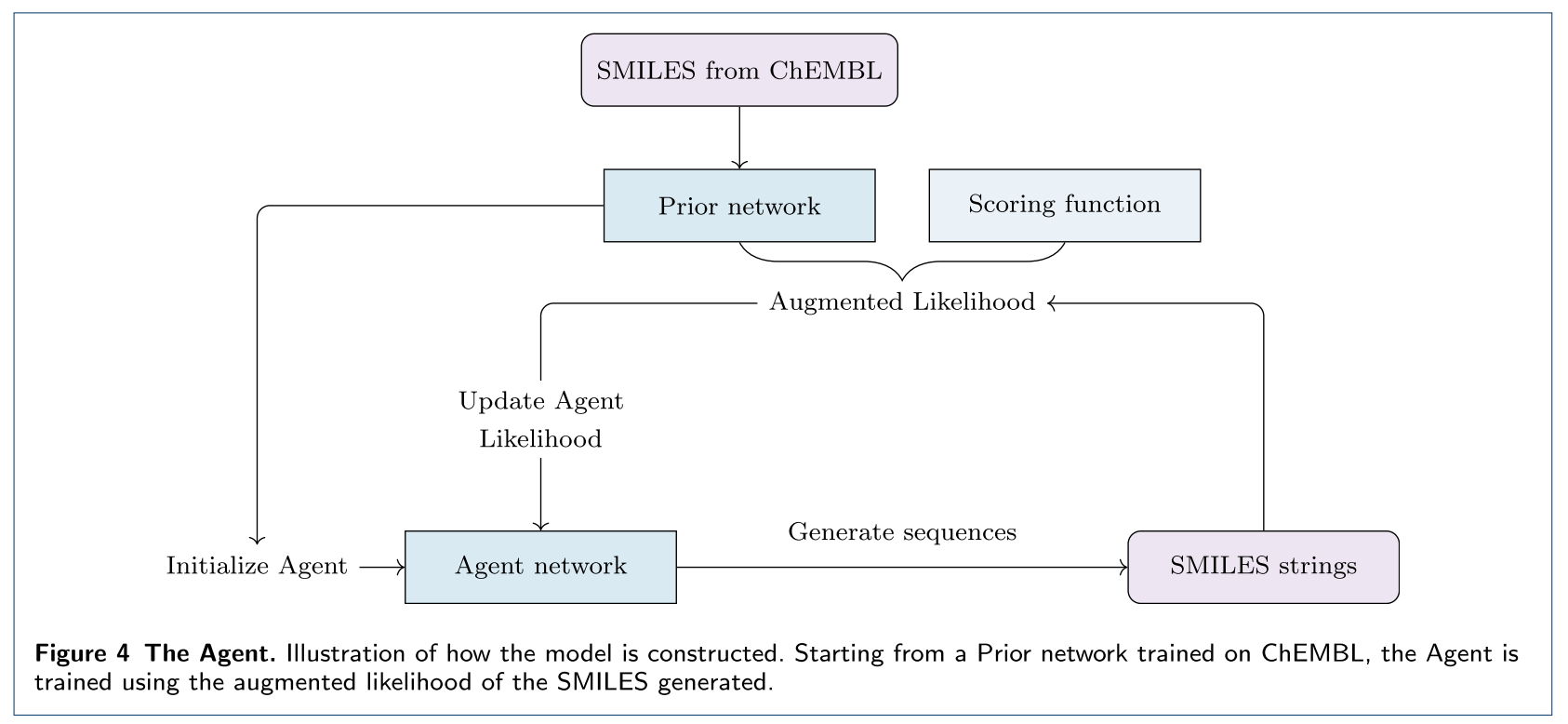
此时可以将生成小分子的过程想象成一场游戏,每生成一个字母就是一个行为(action, \(A= \{ a_1, a_2, .. a_n \}\)),生成完整个小分子就是完成了整场游戏(episode),获得一个“分数”(S(A))。优化的目的是尽可能让生成的小分子拥有较高的分数。比如这里的分数可以是“与某个分子的相似程”。 于是得到一个小分子的概率就可以描述成:
$$ P(A) = \prod_{t=1}^{T}\pi (a_t|s_t) $$
而具体损失函数如下得到:
首先我们要在Prior模型的基础上增加打分,成为“目标概率”,即:
$$ logP(A)_U=logP(A)_{Prior}+\sigma S(A) $$
损失函数则要让Agent做这个决策的概率接近这个目标概率,即:
$$ L(\Theta) = [logP(A)_U-logP(A)_A]^2 $$
源码及使用方法
源码和使用方法见:https://github.com/MarcusOlivecrona/REINVENT
无需REINVENT,只需要搭建环境然后直接使用即可,环境包括:1
2
3
4
5Python 3.6
PyTorch 0.1.12
RDkit
Scikit-Learn (for QSAR scoring function)
tqdm (for training Prior)
安装方法:1
conda install -c rdkit rdkit scikit-learn tqdm pytorch=0.1.12 python=3.6
另外如果发现缺少其他包(比如pexpect),建议直接用pip安装即可,附笔者的pip环境:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27bokeh==1.0.1
certifi==2018.4.16
cffi==1.11.5
cycler==0.10.0
Jinja2==2.10
kiwisolver==1.0.1
MarkupSafe==1.1.0
matplotlib==2.2.2
numpy==1.14.3
olefile==0.45.1
packaging==18.0
pandas==0.20.3
pexpect==4.6.0
Pillow==5.1.0
ptyprocess==0.6.0
pycparser==2.18
pyparsing==2.2.0
python-dateutil==2.6.1
pytz==2017.2
PyYAML==3.13
scikit-learn==0.19.1
scipy==1.1.0
six==1.11.0
torch==0.4.0
torchvision==0.2.1
tornado==5.0.2
tqdm==4.15.0
环境搭配完后输入:./main.py --scoring-function activity_model --num-steps 10即可看到生成的分子,
其中activity_model是原作者预设的打分函数,可以根据自己需要修改,也可以使用原作者预设的相似性打分函数,详见score_functions.py。
原作者已经使用 ChEMBL 训练过了 Prior 模型,如果想自己训练,则根据 GitHub 教程,先准备好分子文件mol.smi,然后输入./data_structs.py mols.smi,再输入./train_prior.py,笔者并未尝试,不知要训练多久。
有描述不对的地方欢迎留言反馈
参考文献
(1) Olivecrona, M.; Blaschke, T.; Engkvist, O.; Chen, H. Molecular De Novo Design through Deep Reinforcement Learning. 2017. http://arxiv.org/abs/1704.07555