Python 中用List与Dict代替树,由AtomSignature到Smiles的转换脚本
Comment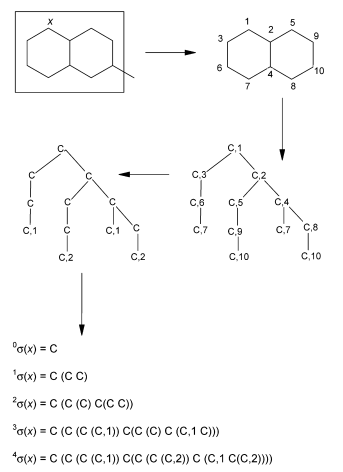
CDK(Chemistry Develop Kit)中有一个功能就是由分子转化成原子信息(AtomSignaure,下文简称AS),其实就是以树的方式描述原子之间的关系。
可是这样得到的原子信息没有办法直接利用(至少我不知道有什么软件或者脚本可以实现,网上并没有查到)。于是今天花了一天的时间,自己用手用Python实现了从AS到SMIELS的转换。
想要转换,先要搞懂AS的表达形式是什么,如图1可知,它可以理解为,以某个碳原子为起点,即树的根,寻找与碳原子相邻的原子作为根节点的一个子节点,以此类推。如果碰到环的情况,与SMILES类似,用[C,x]。x是数字,表示它们指的是同一个碳原子,且一般不会再有子节点。然后是字符串表达形式,显然是根据深度优先的遍历顺序,深度每增加1,则用左括号表示,深度倒退1,则用右括号表示。
这里的括号与SMILES有相同点与不同点。相同点是都是表示“分支”,但是SMILES中括号是支链的意思,而主链则不需要用括号,如果把括号中内容全部去掉,则只剩下一条主链。而AS中括号表示的是子节点,可以这样理解,C1(C2C3);C2=(C5C4)以此迭代下去,并没有主链的存在。所以显然SMILES与AS的表达方式是不同的,不可以简单的取代。
我使用的方法是将AS的线性表达先转化成树,然后再由树转化成SMILES式。
我使用的是Knime下的CDK插件,生成的AS格式如下:
C)[N]pN))
下文是针对这么一个字符串让它最终变成SMILES的格式:
1. AS——>树
Python好像有提供操作树的类,但是我个人比较喜欢List与Dict两个数据类型,也是因为这两个数据类型爱上Python的。于是我试着用这两种数据类型来描述一棵树。
很简单,就是树具有根节点和叶子节点,有点像字典的key和value,于是可以这样表达:{C1:[C2,C3]},其中C2 = {C2:[C5,C4]},以此迭代,这样既储存了每个节点的信息,也能很好的具有迭代的性质:键对应的是根节点,值是子节点的数组,子节点本身也是一个字典。
AS本身是以字符串表达,所以要对字符串逐字分析:
每个节点可以看成C的字符串,所以第一步是确定节点信息,即[C],确定方法是当第一次出现”(“表示要进入子节点,也就意味着根节点信息保存完毕,然后根据左括号数等于右括号数,找到对应的右括号,子节点信息保存完毕,进入下一个并列的兄弟节点。树的第一层是[C],对应的子节点信息是:pC)[N]pN)
子节点信息其实就是“C1C2”的形式,也就是出现第一个左括号时表示子节点信息开始,找到对应的右括号后则变成C2即相邻节点的信息。子节点即第三层,继续迭代即可。
所以我把这个过程看成两个部分:寻找当前层所有节点的信息,作为“键”;得到子节点组的信息,进一步迭代。
需要注意的是,像[N]p[N]这样,由于[N]本身就是叶子节点,所以虽然没有括号,但它与p[N]是两个原子,需要作为两个节点处理,如果像前面说的,碰到括号才理解为下一个节点是不行的,我这里利用“]”作为让原子与原子隔开的标记,详见脚本。
碰到没有子节点的时候,理论上也可以认为是一个字典,字典只有键没有值,比如{[C]:},但我个人爱好是直接用字符串表达,就直接是”[C]”,这样的好处第2部分里可以发现。(也不一定是好处,时间有限,我没尝试用字典表达叶子节点)
最终得到字符串List:[{‘[C]’: [{‘p[C]’: [{‘p[c]’: [‘p[C,0]’]}]}, ‘[N]’,{‘p[N]’: [{‘p[C]’: [‘p[C,0]’, ‘p[N]’]}]}]}]”
2. 树——>SMILES
这个分为两步,一步是让非线性的字典表达式转化成线性的字符串;第二步是优化,因为AS的表达与SMILES不同,所以要修改为SMILES样式。
第一步类似于上一步的逆运算,也是需要迭代的。
我把节点线性化分成两种情况,第一种是如果节点只有一个子节点,那么认为它是主链,即没有括号,如果有不止一个子节点,那么最后一个节点为主链,其他为支链需要加括号。之所以是最后一个节点为主链,因为SMILES式中支链是写在主链前面的,如2甲基戊烷可以写成:C1(C2C3)C4C5C6,C2C3是支链,C4C5C6是主链,可见支链写在主链的前面。
如果是叶子节点,则直接返回字符串内容(这需要用到type命令进行类型判定),如果不是,则需要进一步迭代。
这样最终得到的是p[C]这种形式,显然不行,p[C]表示芳香C,但在SMILES中一般表示为c,所以要进一步优化。这种优化还简单,更难的是环的问题,比如环己烷SMILES中环表示的是C1CCCCC1,一共六个碳,但是AS的表达则是(假设从3号碳原子出发)C)C)),能发现会多一个碳原子,因为[C,1]是重复出现的(仔细观察图1也能看出来)所以我们要去掉这个但又要保留“1”这个信号。我们不能直接去掉[C,1]改成c1和1,还要考虑如果第二个C1在支链上,则可能会出现C1CC(1)C这种情况。那么要去掉这个支链信号,如果在主链上,可能会出现C1CC(C)1这种情况,我们要讲1提到前面那个碳中,因为想要表达的是第一个碳和第3个碳相连,具体解决方法可以参考附件脚本。
通过这么个脚本的制作,还能回顾一些文本处理的方法,主要就是Re模块的正则表达式方法。
需要注意的一点,我在编写过程中犯了一个小错误,很有意思:
通过re.findall找到了[C,1],然后通过re.sub改成C1,都没问题,接着我写了类似于下面的代码
Rel = ‘C1’
Ori = ‘[C,1]’
re.sub(Ori,Rel,smiles)
结果就错了。原因是Ori参数写的应该是正则表达式,而不是原字符串,所以’[C,1]’在这里指的是C或1,而不是这段字符串。后来我该成了smiles = smiles.replace(Ori,Rel) 就可以了。